动植物全基因组重测序是对已知基因组序列的物种进行DNA测序,并在此基础上完成个体或群体分析。通过序列比对,可以检测到大量变异信息,包括单核苷酸多态性位点(SNP),插入缺失位点(Insertion/Deletion, InDel)、结构变异(Structure Variation, SV)位点,拷贝数变异(Copy Number-Variation, CNV)位点等,获得同一物种不同个体的遗传变异图谱。利用全基因组重测序技术有助于快速发现与动植物重要性状相关的遗传变异,应用于分子育种中,缩短育种周期。

滚环扩增构建DNB测序文库,PCR-free重测序检测lnDel更精准,无index hopping之忧, 低dup rate无需人为干预。
DNBSEQ-T20超高通量,
周期短、性价比高。
产品应用

目标性状基因挖掘
群体遗传学研究

变异图谱构建

分子标记开发及
辅助选择育种

物种/品种鉴定
动植物核心
资源普查
技术流程
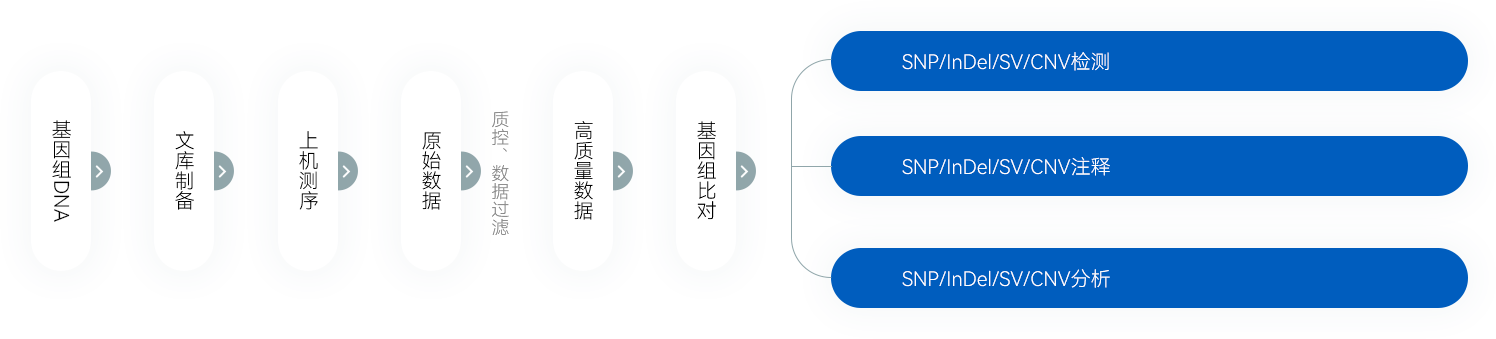
技术参数

样品要求
基因组DNA,无降解或轻微降解

样品需求量(单次)
≥1ug

样品浓度
≥12.5ng/uL

推荐数据量
个体重测序推荐30X以上,群体重测序推荐10X以上