3K水稻重测序&泛基因组研究
研究背景
3,010份水稻(来自全球89个国家和地区)代表了全球78万份水稻种质约95%多样性的核心种质。通过全基因组重测序,每个样本平均测序深度14X,利用重测序数据共检测到32Mb的高质量SNPs和InDels。对亚洲栽培稻群体的结构和分化进行了更为细致和准确的描述和划分,由传统的5个群体增加到9个,分别是东亚(中国)的袖稻、南亚的袖稻、东南亚的袖稻和现代袖稻品种等4个袖稻群体,东南亚的温带粳稻、热带粳稻、亚热带粳稻等3个粳稻群体、以及来自印度和孟加拉的Aus和香稻。
研究首次揭示了亚洲栽培稻品种间存在的大量微细结构(>100bp)变异(SVs,包括易位、缺失、倒位和重复)。着重研究453个测序深度>20X的品系的SVs,利用SVs构建的进化树与SNP构建的进化树类似。大量的SVs可能是不同程度杂种不育和XI与GJ杂种衰退的遗传基础。同时构建了亚洲栽培稻的泛基因组,包括12,770个(62.1%)核心(core)基因家族和9,050个(37.9%)分散式(distributed)基因家族。发现了1.2万个全长新基因和数千个不完整的新基因。核心基因比较古老,大多数的新基因表现更年轻和长度偏短。
研究策略
最初测序3,024份水稻样本,后来进行质控过滤掉14份,最终保留3,010份水稻样本进行深度研究。3K RG测序数据比对到参考基因组日本晴Nipponbare上检泌SNPs、InDels。合并Nipponbare基因组序列和无冗余的新组装的基因组序列构建泛基因组。利用测序深度>20X,比对深度>15X的453个水稻材料进行SVs和PAVs分析。
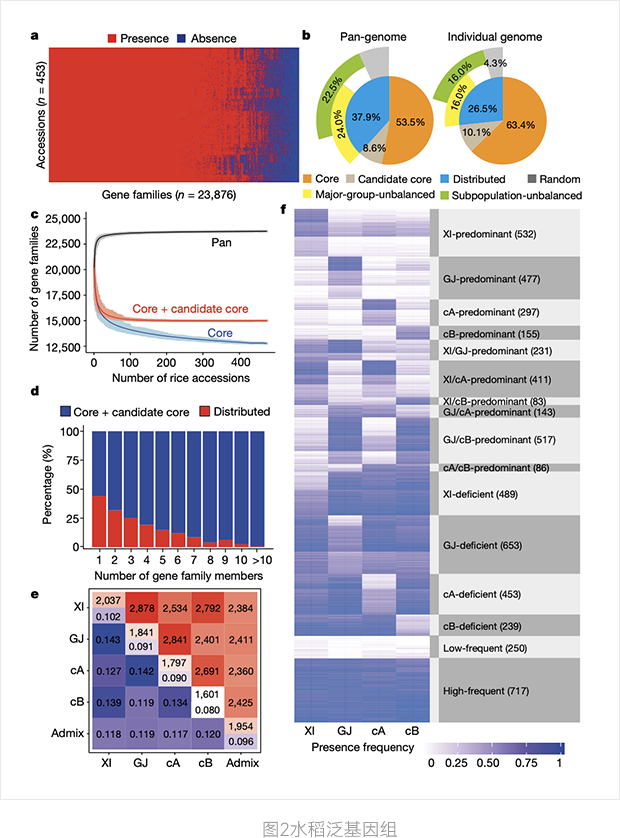
a. 基因家族PAVs
b. 泛基因组和一个单独旳基因组旳组成成份
c. 基于500个随机筛选旳水稻基因组模拟泛 基因组和核心基因组
d. 核心和分散式基因家族比例
e. 两个品系间基因家族平均数量差异
f. 5733主要群组不平衡基因家族特性
研究结果
研究结果
参考文献
Wang W, Mauleon R, Hu Z, et al. Genomic variation in 3,010 diverse accessions of Asian cultivated rice[J]. Nature, 2018,557(7703): 43.