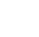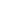育种多组学 生信分析
智数生物依托高通量测序技术和专业的生物信息学分析团队,精心打造了一系列尖端的针对基因组、转录组和表型组数据的分析工具与流程,挖掘大数据背后的生物学意义和实际应用价值。
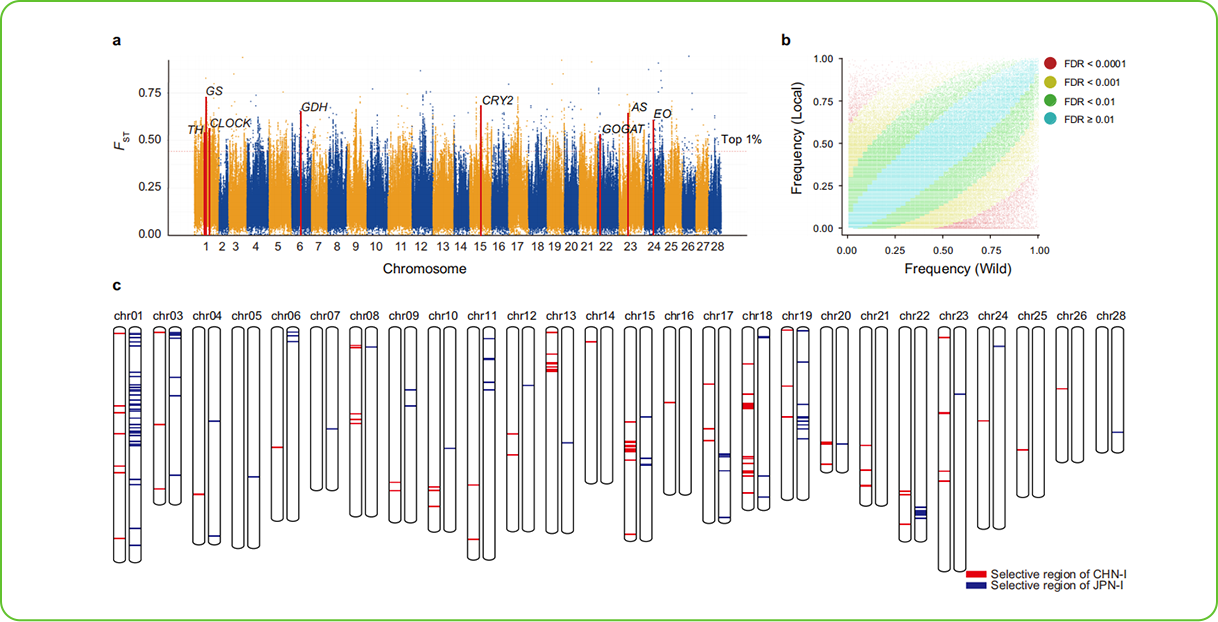
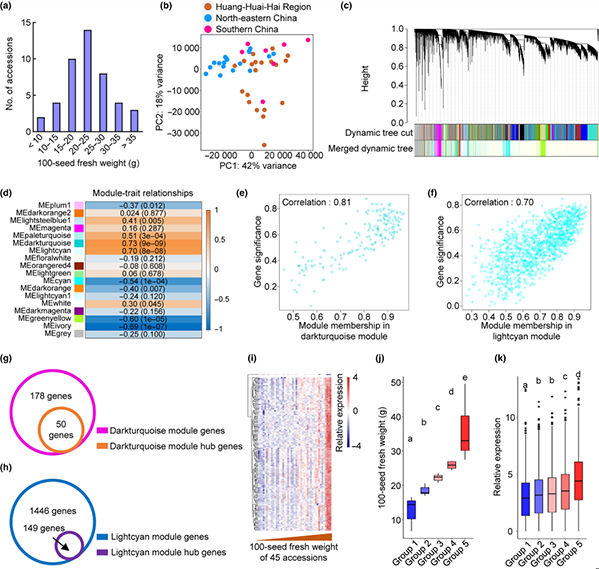
通过对群体或个体多组学数据进行分析,揭示不同个体基因组存在的遗传变异和重要生物学过程的分子调控机制,评估群体结构和遗传多样性,获得与目标性状关联的基因和分子标记。
通过对群体或个体多组学数据进行分析,揭示不同个体基因组存在的遗传变异和重要生物学过程的分子调控机制,评估群体结构和遗传多样性,获得与目标性状关联的基因和分子标记。
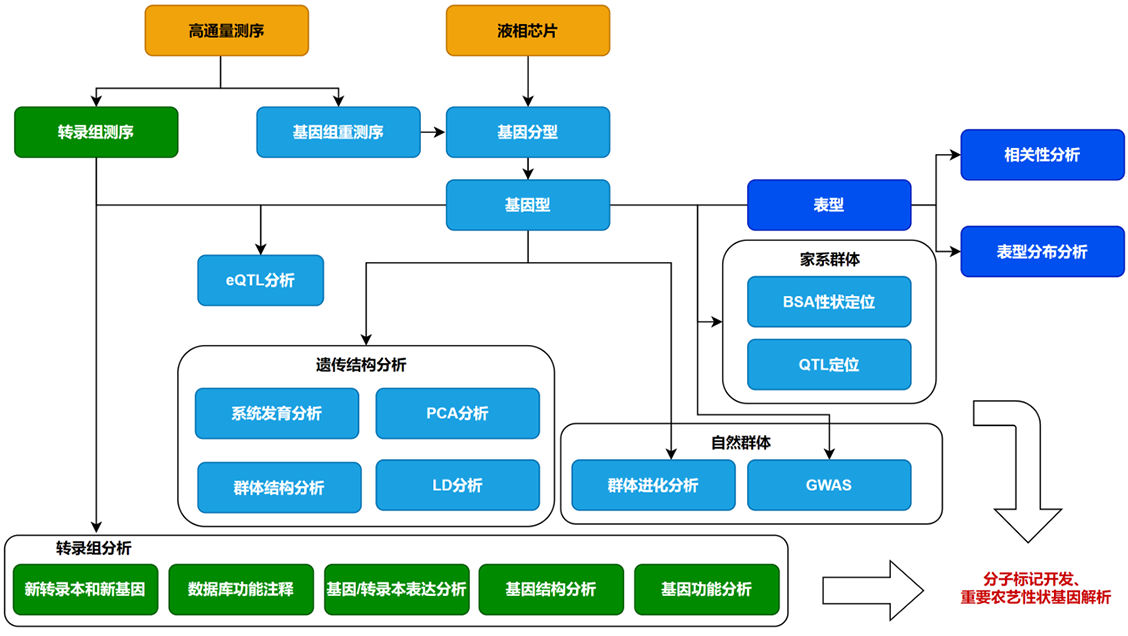

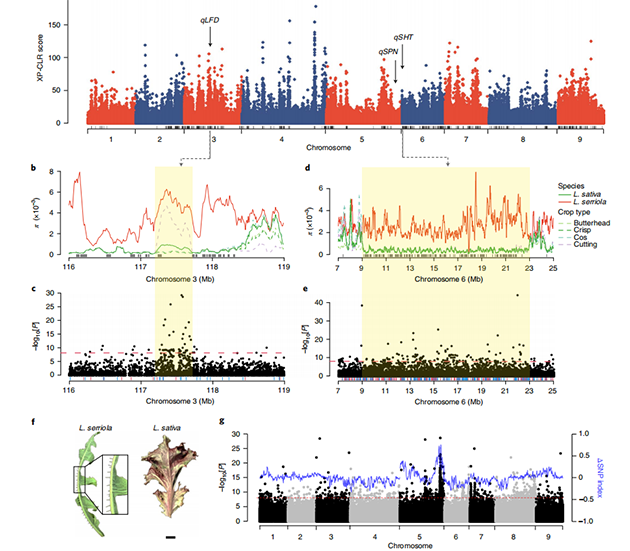
基于华大测序平台BGISEQ-500对445份栽培生菜及其野生近缘种进行测序,以研究它们的系统发育关系和种群结构。
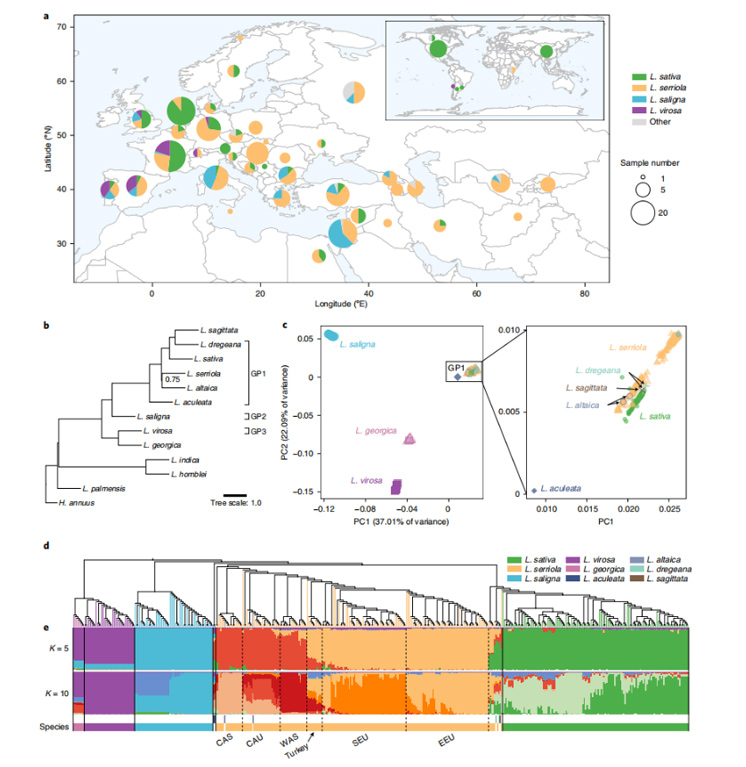
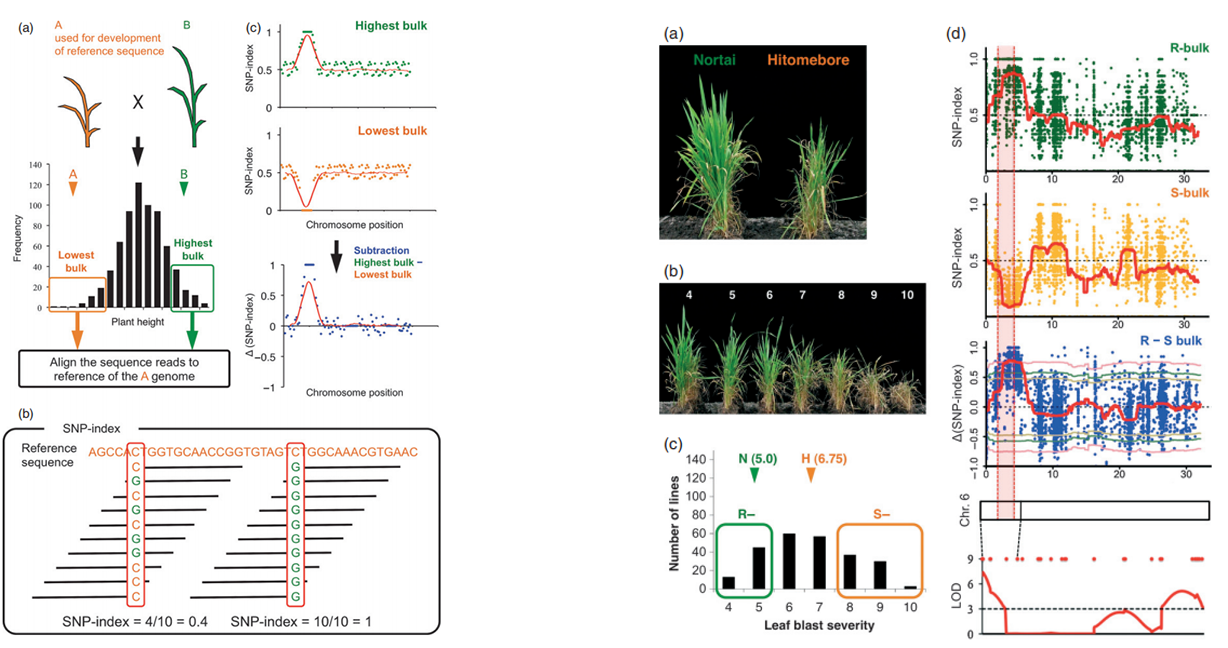
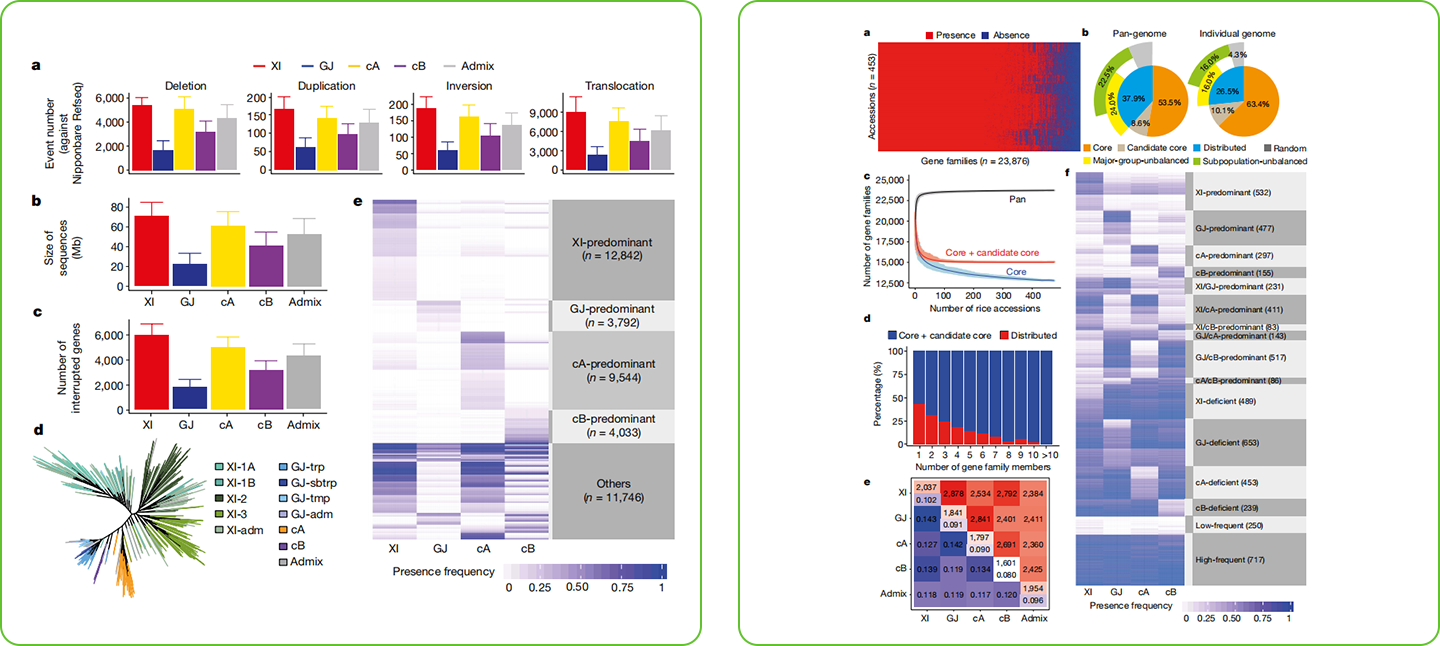
在3,010份水稻的研究中,Wang等人对453份高覆盖率水稻材料的SVs(结构变异)进行了总结。
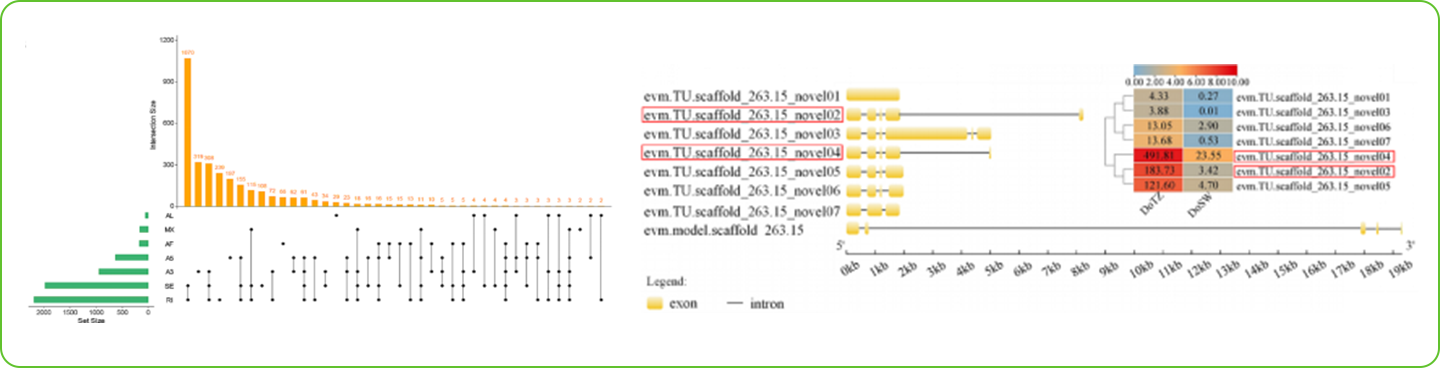
采用了一种“map-to-pan”的策略来构建了水稻的泛基因组。
Wang, W., Mauleon, R., Hu, Z., Chebotarov, D., Tai, S. et al. Genomic variation in 3,010 diverse accessions of Asian cultivated rice. Nature, 2018, 557: 43–49.

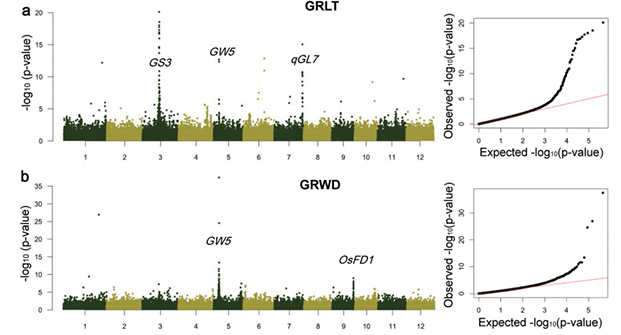
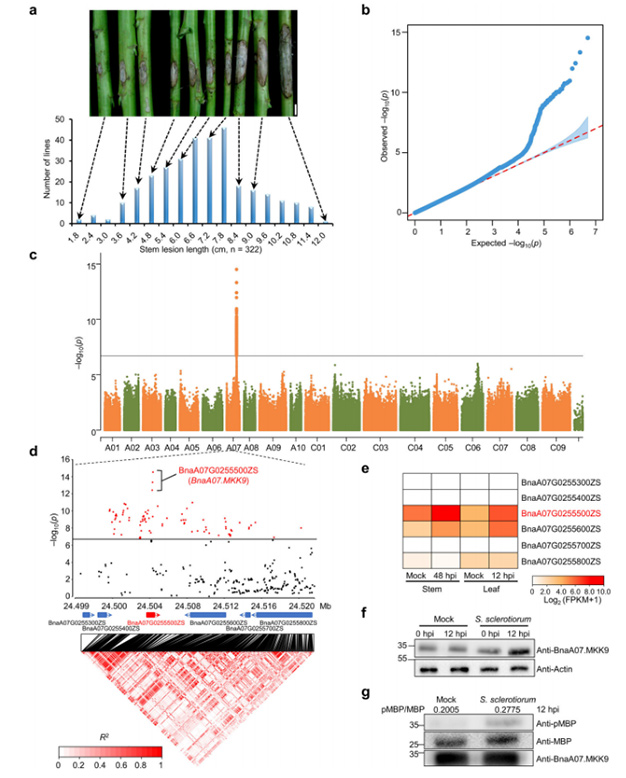
对218个水稻重组自交系(RILs)群体进行测序,分析得到水稻基因组各染色体上SNP的热图分布,定位了控制株高的数量性状位点(QTL),绘制与产量相关的性状的QTL在染色体上的位置分布。
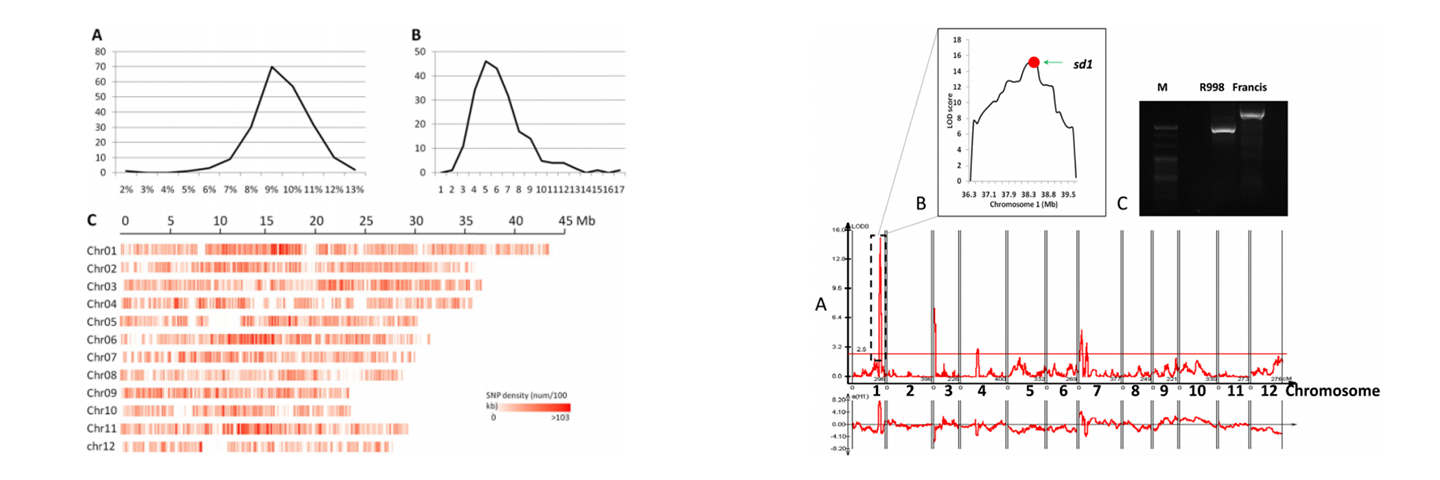
ZhuM, M., Liu, D., Liu, W., Li, D., Liao Y. et al. QTL mapping using an ultra-high-density SNP map reveals a major locus for grain yield in an elite rice restorer R998. Sci Rep. 2017, 7(1): 10914.